Scopri come utilizzare gli strumenti OCR, Apache Spark e altri componenti Apache Hadoop per elaborare immagini PDF su larga scala.
Le tecnologie di riconoscimento ottico dei caratteri (OCR) sono notevolmente avanzate negli ultimi 20 anni. Tuttavia, durante quel periodo, c'è stato uno sforzo minimo o nullo per coniugare l'OCR con architetture distribuite come Apache Hadoop per elaborare un gran numero di immagini in tempo quasi reale.
In questo post imparerai come utilizzare strumenti open source standard insieme a componenti Hadoop come Apache Spark, Apache Solr e Apache HBase per fare proprio questo per un caso d'uso di informazioni sui dispositivi medici. In particolare, utilizzerai un set di dati pubblico per convertire il testo narrativo in campi ricercabili.
Sebbene questo esempio si concentri sulle informazioni sui dispositivi medici, può essere applicato in molti altri scenari in cui è richiesta l'elaborazione e la persistenza delle immagini. Le compagnie di assicurazione, ad esempio, possono rendere ricercabili tutti i loro documenti scansionati nei file dei sinistri per una migliore risoluzione dei sinistri. Allo stesso modo, il dipartimento della catena di approvvigionamento in un impianto di produzione potrebbe scansionare tutte le schede tecniche dei fornitori di parti e renderle ricercabili dagli analisti.
Caso d'uso:registrazione del dispositivo medico
Gli ultimi anni hanno visto una serie di cambiamenti nel campo della registrazione elettronica dei prodotti farmaceutici. Lo standard ISO IDMP (Identificazione dei prodotti medici) è uno di questi formati di messaggio per la registrazione dei prodotti e delle sostanze in essi contenute, con l'ID del medicinale, l'ID dell'imballaggio e l'ID del lotto utilizzati per tracciare i prodotti in caso di esperienze avverse, illegale importazione, contraffazione e altri problemi di farmacovigilanza. La norma richiede che non solo i nuovi prodotti debbano essere registrati, ma che anche la scheda più vecchia/archiviata di ogni prodotto a cui il pubblico potrebbe essere esposto debba essere fornita in forma elettronica.
Per conformarsi agli standard IDMP in diverse aziende, le aziende devono essere in grado di estrarre ed elaborare dati da più origini dati, come RDBMS e, in alcuni casi, schede tecniche di prodotto legacy. Sebbene sia noto come acquisire dati da RDBMS tramite tecnologie come Apache Sqoop, l'elaborazione dei documenti legacy richiede un po' più di lavoro. Per la maggior parte, i documenti devono essere inseriti e il testo pertinente deve essere estratto programmaticamente su larga scala utilizzando le tecnologie OCR esistenti.
Set di dati
Utilizzeremo un set di dati della FDA che contiene tutti i 510(k) documenti presentati dai produttori di dispositivi medici dal 1976. La sezione 510(k) del Food, Drug and Cosmetic Act richiede ai produttori di dispositivi che devono registrarsi di notificare FDA della loro intenzione di commercializzare un dispositivo medico con almeno 90 giorni di anticipo.
Questo set di dati è utile per diversi motivi in questo caso:
- I dati sono gratuiti e di pubblico dominio.
- I dati si adattano perfettamente al regolamento europeo, che entrerà in vigore a luglio 2016 (dove i produttori devono attenersi ai nuovi standard di dati). Le otturazioni della FDA contengono importanti informazioni rilevanti per ottenere una visione completa dell'IDMP.
- Il formato dei documenti (PDF) ci consente di dimostrare tecniche OCR semplici ma efficaci quando si tratta di documenti di più formati.
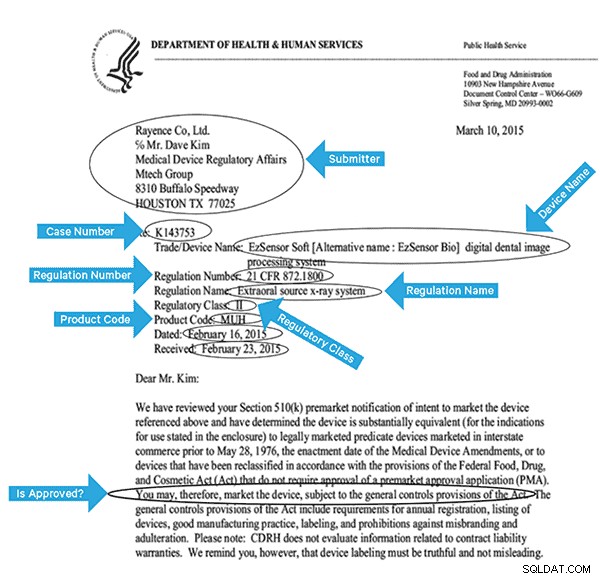
Per indicizzare efficacemente questi dati, dovremo estrarre alcuni campi dalle immagini. Di seguito è riportato un documento di esempio, con i potenziali campi che possono essere estratti.
Architettura di alto livello
Per questo caso d'uso, i PDF vengono archiviati in HDFS ed elaborati utilizzando le librerie Spark e OCR. (Il passaggio di importazione non rientra nell'ambito di questo post, ma potrebbe essere semplice come eseguire hdfs -dfs -put o utilizzando un'interfaccia webhdfs.) Spark consente l'uso di codice quasi identico in un'applicazione Spark Streaming per lo streaming quasi in tempo reale e HBase è un supporto di archiviazione perfetto per l'accesso casuale a bassa latenza ed è adatto per archiviare immagini, con la nuova funzionalità MOB, per l'avvio. Cloudera Search (che si basa su Apache Solr) è l'unica soluzione di ricerca che si integra nativamente con HBase, consentendo così di creare indici secondari.
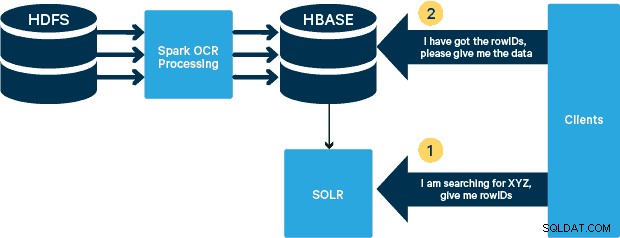
Impostazione del tavolo dei dispositivi medici in HBase
Manterremo lo schema per il nostro caso d'uso semplice. L'ID riga sarà il nome del file e ci saranno due famiglie di colonne:"info" e "obj". La famiglia di colonne "info" conterrà tutti i campi che abbiamo estratto dalle immagini. La famiglia di colonne "obj" conterrà i byte dell'oggetto binario effettivo, in questo caso PDF. Il nome della tabella nel nostro caso sarà "mdds".
Sfrutteremo la funzionalità HBase MOB (oggetto medio) introdotta in HBASE-11339. Per configurare HBase per gestire MOB, sono necessari alcuni passaggi aggiuntivi, ma, convenientemente, le istruzioni possono essere trovate a questo link.
Esistono molti modi per creare la tabella in HBase a livello di codice (API Java, API REST o un metodo simile). Qui useremo la shell HBase per creare la tabella "mdds" (usando intenzionalmente un nome di famiglia di colonne descrittivo per rendere le cose più facili da seguire). Vogliamo che la famiglia di colonne "info" venga replicata in Solr, ma non i dati MOB.
Il comando seguente creerà la tabella e consentirà la replica su una famiglia di colonne denominata "info". È fondamentale specificare l'opzione REPLICATION_SCOPE => '1' , altrimenti HBase Lily Indexer non riceverà alcun aggiornamento da HBase. Vogliamo utilizzare il percorso MOB in HBase per oggetti più grandi di 10 MB. A tale scopo creiamo anche un'altra famiglia di colonne, denominata "obj", utilizzando i seguenti parametri per i MOB:
IS_MOB => true, MOB_THRESHOLD => 10240000
Il IS_MOB il parametro specifica se questa famiglia di colonne può memorizzare MOB, mentre MOB_THRESHOLD specifica dopo quanto grande deve essere l'oggetto per essere considerato un MOB. Quindi, creiamo la tabella:
create 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000}
Per confermare che la tabella è stata creata correttamente, eseguire il seguente comando nella shell HBase:
hbase(main):001:0> describe 'mdds'
Table mdds is ENABLED
mdds
COLUMN FAMILIES DESCRIPTION
{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1', VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'FOREVER', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
2 row(s) in 0.3440 seconds
Elaborazione delle immagini scansionate con Tesseract
L'OCR ha fatto molta strada in termini di gestione delle variazioni dei caratteri, del rumore dell'immagine e dei problemi di allineamento. Qui utilizzeremo il motore OCR open source Tesseract, originariamente sviluppato come software proprietario presso i laboratori HP. Da allora lo sviluppo di Tesseract è stato rilasciato come software open source ed è stato sponsorizzato da Google dal 2006.
Tesseract è una libreria software altamente portatile. Utilizza la libreria di elaborazione delle immagini Leptonica per generare un'immagine binaria eseguendo la soglia adattiva su un'immagine grigia o colorata.
L'elaborazione segue una tradizionale pipeline passo-passo. Di seguito è riportato il flusso approssimativo dei passaggi:
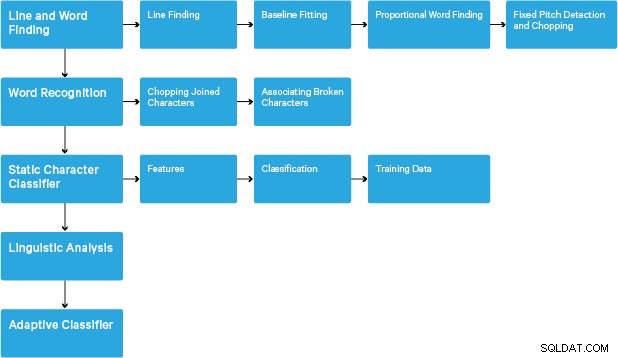
L'elaborazione inizia con un'analisi dei componenti collegati, che si traduce nella memorizzazione dei componenti trovati. Questo passaggio aiuta nell'ispezione dell'annidamento dei contorni e del numero di contorni figlio e nipote.
In questa fase, i contorni vengono raccolti insieme, semplicemente nidificando, in Binary Large Objects (BLOB). I BLOB sono organizzati in righe di testo e le righe e le aree vengono analizzate per il testo a passo fisso o proporzionale. Le righe di testo sono suddivise in parole in modo diverso a seconda del tipo di spaziatura dei caratteri. Il testo a passo fisso viene tagliato immediatamente dalle celle dei caratteri. Il testo proporzionale è suddiviso in parole utilizzando spazi definiti e spazi sfocati.
Il riconoscimento procede quindi come un processo a due passaggi. Nel primo passaggio, si tenta di riconoscere ogni parola a turno. Ogni parola soddisfacente viene passata a un classificatore adattivo come dati di addestramento. Il classificatore adattivo ha quindi la possibilità di riconoscere in modo più accurato il testo in basso nella pagina. Poiché il classificatore adattivo potrebbe aver appreso qualcosa di utile troppo tardi per dare un contributo vicino alla parte superiore della pagina, viene eseguito un secondo passaggio sulla pagina, in cui le parole che non sono state riconosciute abbastanza bene vengono nuovamente riconosciute. Un'ultima fase risolve gli spazi sfocati e verifica le ipotesi alternative per l'altezza x per individuare il testo in maiuscolo.
Tesseract nella sua forma attuale è completamente compatibile con Unicode e addestrato per diverse lingue. Sulla base della nostra ricerca, è una delle librerie open source più accurate disponibili per l'OCR. Come accennato in precedenza, Tesseract usa Leptonica. Utilizziamo anche Ghostscript per dividere i file PDF in immagini. (Puoi dividere in un formato di compressione delle immagini a tua scelta; abbiamo scelto PNG.) Queste tre librerie sono scritte in C++ e per invocarle dai programmi Java/Scala, dobbiamo utilizzare le implementazioni delle corrispondenti interfacce native Java. Nel nostro lavoro, utilizziamo i collegamenti JNI di JavaPresets. (Le istruzioni di compilazione sono disponibili di seguito.) Abbiamo usato Scala per scrivere il driver Spark.
val renderer :SimpleRenderer = new SimpleRenderer( ) renderer.setResolution( 300 ) val images:List[Image] = renderer.render( document )
Leptonica legge le immagini divise dal passaggio precedente.
ImageIO.write(
x.asInstanceOf[RenderedImage],
"png",
imageByteStream
)
val pix: PIX = pixReadMem (
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ),
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacity( )
) Usiamo quindi le chiamate API Tesseract per estrarre il testo. Assumiamo che i documenti siano in inglese qui, quindi il secondo parametro del metodo Init è "eng".
val api: TessBaseAPI = new TessBaseAPI( ) api.Init( null, "eng" ) api.SetImage(pix) api.GetUTF8Text().getString()
Dopo che le immagini sono state elaborate, estraiamo alcuni campi dal testo e li inviamo a HBase.
def populateHbase (
fileName:String,
lines: String,
pdf:org.apache.spark.input.PortableDataStream) : Unit =
{
/** Configure and open a HBase connection */
val mddsTbl = _conn.getTable( TableName.valueOf( "mdds" ));
val cf = "info"
val put = new Put( Bytes.toBytes( fileName ))
/**
* Extract Fields here using Regexes
* Create Put objects and send to HBase
*/
val aAndCP = """(?s)(?m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe: (\w\d\d\d\d\d\d).*""".r
……..
lines match {
case
aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "submitter_info" ),
Bytes.toBytes( addr ) ).add( Bytes.toBytes( cf ),
Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum ))
case _ => println( "did not match a regex" )
}
…….
lines.split("\n").foreach {
val regNumRegex = """Regulation Number:\s+(.+)""".r
val regNameRegex = """Regulation Name:\s+(.+)""".r
……..
…….
_ match {
case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "reg_num" ),
…….
…..
case _ => print( "" )
}
}
put.add( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines ))
val pdfBytes = pdf.toArray.clone
put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( "pdf" ), pdfBytes )
mddsTbl.put( put )
…….
}
Se osservi attentamente il codice sopra, subito prima di inviare l'oggetto Put a HBase, inseriamo i byte PDF non elaborati nella famiglia di colonne "obj" della tabella. Usiamo HBase come livello di archiviazione per i campi estratti e per l'immagine grezza. Ciò rende veloce e conveniente per l'applicazione estrarre l'immagine originale, se necessario. Il codice completo può essere trovato qui. (Vale la pena notare che mentre abbiamo utilizzato API HBase standard per creare oggetti Put per HBase, in un sistema di produzione reale, sarebbe saggio considerare l'utilizzo di API SparkOnHBase, che consentono aggiornamenti batch di HBase da Spark RDD.)
Pipeline di esecuzione
Siamo stati in grado di elaborare ogni PDF in una struttura seriale. Per ridimensionare l'elaborazione, abbiamo scelto di elaborare questi PDF in modo distribuito utilizzando Spark. Il grafico seguente mostra come combiniamo diverse fasi di questa elaborazione per trasformare il flusso di lavoro in una semplice chiamata macro da Spark e caricare i dati in HBase.
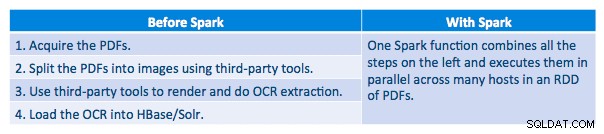
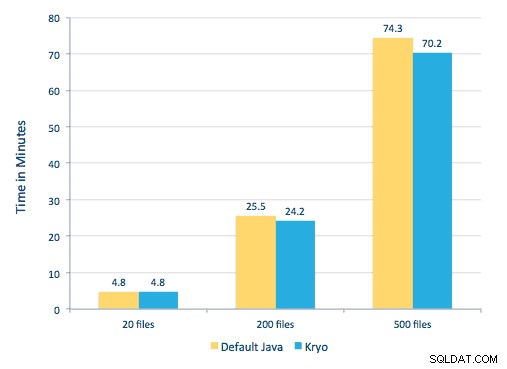
Abbiamo anche provato a fare un confronto tra i metodi di serializzazione, ma, con il nostro set di dati, non abbiamo riscontrato differenze significative nelle prestazioni.
Configurazione ambiente
Hardware utilizzato:cluster a cinque nodi con 15 GB di memoria, 4 vCPU e 2 SSD da 40 GB
Dato che stavamo usando le librerie C++ per l'elaborazione, abbiamo usato i collegamenti JNI che possono essere trovati qui.
Crea i collegamenti JNI per Tesseract e Leptonica dai predefiniti javaCPP:
-
- Su tutti i nodi:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets- Compila Leptonica.
cd leptonica ./cppbuild.sh install leptonica cd cppbuild/linux-x86_64/leptonica-1.72/ LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && sudo make install cd ../../../ mvn clean install cd ..
- Costruisci Tesseract.
- Su tutti i nodi:
cd tesseract ./cppbuild.sh install tesseract cd tesseract/cppbuild/linux-x86_64/tesseract-3.03 LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && make install cd ../../../ mvn clean install cd ..
- Crea preset javaCPP.
mvn clean install --projects leptonica,tesseract
Usiamo Ghostscript per estrarre le immagini dai PDF. Le istruzioni per costruire Ghostscript, corrispondenti alle versioni di Tesseract e Leptonica usate qui, sono le seguenti. (Assicurati che Ghostscript non sia installato nel sistema tramite il gestore pacchetti.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gz tar zxvf ghostscript-9.16.tar.gz cd ghostscript-9.16 ./autogen.sh && ./configure --prefix=/usr --disable-compile-inits --enable-dynamic sudo make && make soinstall && install -v -m644 base/*.h /usr/include/ghostscript && ln -v -s ghostscript /usr/include/ps (Depending on your ldpath setting, you may have to do) : sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Assicurati che tutte le librerie necessarie siano nel percorso di classe. Mettiamo tutti i jar rilevanti in una directory chiamata lib. La virgola è importante di seguito:
$ for i in `ls lib/*`; do export MY_JARS=./$i,$MY_JARS; done tesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Invochiamo il programma Spark come segue. Dobbiamo specificare extraLibraryPath per le librerie Ghostscript native; l'altra conf è necessaria per Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor- cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --conf spark.executorEnv.TESSDATA_PREFIX=/home/vsingh/javacpp- presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --conf spark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/ lib/htrace-core-3.1.0-incubating.jar --driver-class-path /etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0- incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer --conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Creazione di una collezione Solr
Solr si integra perfettamente con HBase tramite Lily HBase Indexer. Per capire come viene eseguita l'integrazione dell'integrazione di Lily Indexer con HBase, puoi rispolverare il nostro post precedente nella sezione "Capire la replica di HBase e Lily HBase Indexer".
Di seguito descriviamo i passaggi che devono essere eseguiti per creare gli indici:
- Genera un file di configurazione schema.xml di esempio:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Modifica il file schema.xml in
$HOME/solrcfg, specificando i campi di cui abbiamo bisogno per la nostra raccolta. Il file completo può essere trovato qui. - Carica le configurazioni Solr su ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Genera la raccolta Solr con 2 frammenti (-s 2) e 2 repliche (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
Nel comando sopra abbiamo creato una raccolta Solr con due parametri di frammenti (-s 2) e due repliche (-r 2). I parametri erano sufficienti per il nostro corpus, ma in una distribuzione effettiva si dovrebbe impostare il numero sulla base di altre considerazioni al di fuori del nostro ambito di discussione qui.
Registrazione dell'indicizzatore
Questo passaggio è necessario per aggiungere e configurare l'indicizzatore e la replica HBase. Il comando seguente aggiornerà ZooKeeper e aggiungerà mdds_indexer come peer di replica per HBase. Inserisce anche le configurazioni in ZooKeeper, che Lily HBase Indexer utilizzerà per puntare alla raccolta corretta in Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argomenti:
-n mdds_indexer– specifica il nome dell'indicizzatore che verrà registrato in ZooKeeper-c indexer-config.xml– file di configurazione che specificherà il comportamento dell'indicizzatore-cp solr.zk=localhost:2181/solr– specifica la posizione di ZooKeeper e Solr config. Questo dovrebbe essere aggiornato con la posizione specifica dell'ambiente di ZooKeeper.-cp solr.collection=mdds_collection– specifica quale collezione aggiornare. Richiama il passaggio di configurazione Solr in cui abbiamo creato la raccolta1.
Il index-config.xml file è relativamente semplice in questo caso; tutto ciò che fa è specificare all'indicizzatore quale tabella guardare, la classe che verrà utilizzata come mapper (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ) e il percorso del file di configurazione Morphline. Per impostazione predefinita, il tipo di mappatura è impostato su riga , nel qual caso il documento Solr diventa l'intera riga. Param name="morphlineFile" specifica la posizione del file di configurazione Morphlines. La posizione potrebbe essere un percorso assoluto del tuo file Morphlines, ma poiché stai utilizzando Cloudera Manager, specifica il percorso relativo come morphlines.conf.
Il contenuto del file di configurazione di hbase-indexer può essere trovato qui.
Configurazione e avvio di Lily HBase Indexer
Quando si abilita Lily HBase Indexer, è necessario specificare la logica di trasformazione Morphlines che consentirà a questo indicizzatore di analizzare gli aggiornamenti della tabella Dispositivo medico ed estrarre tutti i campi pertinenti. Vai su Servizi e scegli Lily HBase Indexer che hai aggiunto in precedenza. Seleziona Configurazioni->Visualizza e modifica->A livello di servizio->Morfline . Copia e incolla il file Morphlines.
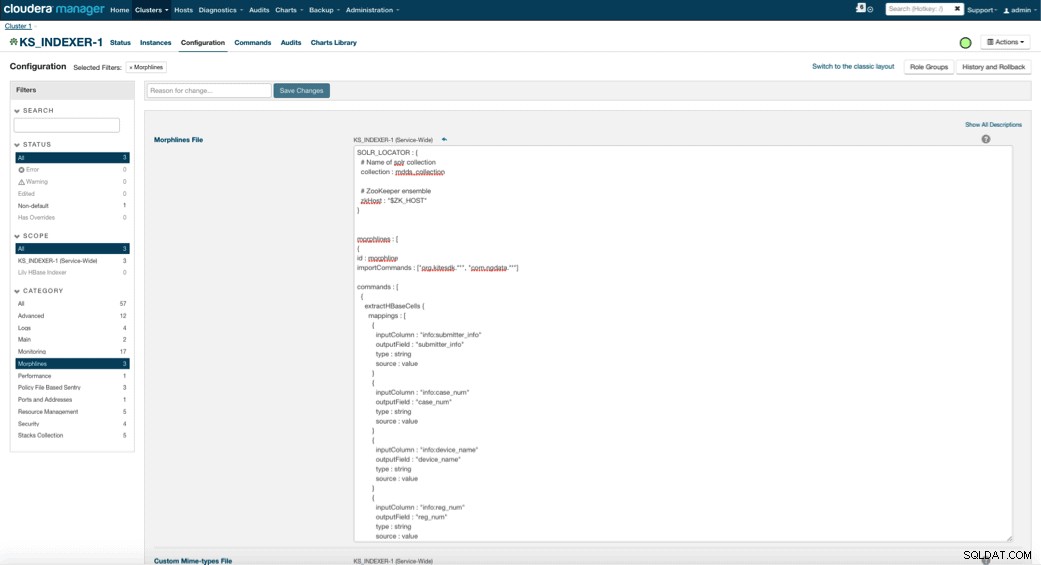
La libreria delle morfoline dei dispositivi medici eseguirà le seguenti azioni:
- Leggi gli eventi email di HBase con
extractHBaseCellscomando - Converti la data/timestamp in un campo che Solr capirà, con il
convertTimestampcomandi - Elimina tutti i campi extra che non abbiamo specificato in schema.xml, con
sanitizeUknownSolrFieldscomando
Scarica una copia di questo file Morphlines da qui.
Una nota importante è che il campo id verrà generato automaticamente da Lily HBase Indexer. Tale impostazione è configurabile nel file index-config.xml sopra specificando l'attributo unique-key-field. È consigliabile lasciare il nome id predefinito, poiché non è stato specificato nel file xml sopra, il campo id predefinito è stato generato e sarà una combinazione di RowID.
Accesso ai dati
Puoi scegliere tra molti strumenti visivi per accedere alle immagini indicizzate. HUE e Solr GUI sono entrambe ottime opzioni. HBase consente anche una serie di tecniche di accesso, non solo da una GUI ma anche tramite la shell HBase, API e persino semplici tecniche di scripting.
L'integrazione con Solr ti offre una grande flessibilità e può anche fornire opzioni di ricerca molto semplici e avanzate per i tuoi dati. Ad esempio, la configurazione del file schema.xml di Solr in modo tale che tutti i campi all'interno dell'oggetto e-mail siano archiviati in Solr consente agli utenti di accedere ai corpi dei messaggi completi tramite una semplice ricerca, con il compromesso di spazio di archiviazione e complessità di calcolo. In alternativa, puoi configurare Solr per memorizzare solo un numero limitato di campi, come l'id. Con questi elementi, gli utenti possono cercare rapidamente Solr e recuperare il rowID che a sua volta può essere utilizzato per recuperare singoli campi o l'intera immagine dalla stessa HBase.
L'esempio sopra memorizza solo l'ID riga in Solr ma gli indici su tutti i campi estratti dall'immagine. La ricerca di Solr in questo scenario recupera gli ID riga HBase, che è quindi possibile utilizzare per interrogare HBase. Questo tipo di configurazione è ideale per Solr in quanto mantiene bassi i costi di archiviazione e sfrutta appieno le capacità di indicizzazione di Solr.
Query di esempio
Di seguito sono riportati alcuni esempi di query che possono essere eseguiti dall'applicazione in Solr. L'idea è che il client interrogherà inizialmente gli indici Solr, restituendo il rowID da HBase. Quindi interroga HBase per il resto dei campi e/o l'immagine grezza originale.
- Dammi tutti i documenti che sono stati archiviati tra le seguenti date:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01-06T23:59:59.999Z TO 2010-02-06T23:59:59.999Z]
- Dammi i documenti che sono stati archiviati sotto il nome normativo dei sistemi a raggi X mobili:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile x-ray system
- Dammi tutti i documenti che sono stati archiviati dai produttori cinesi:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Gli ID dei documenti Solr sono gli ID di riga in HBase; la seconda parte della query sarà indirizzata a HBase per estrarre i dati (compreso il PDF grezzo se richiesto).
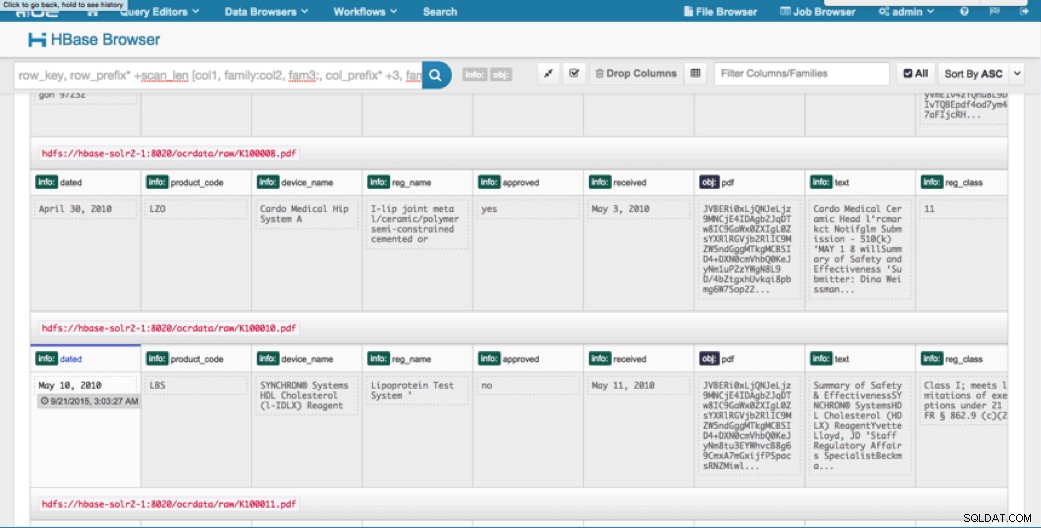
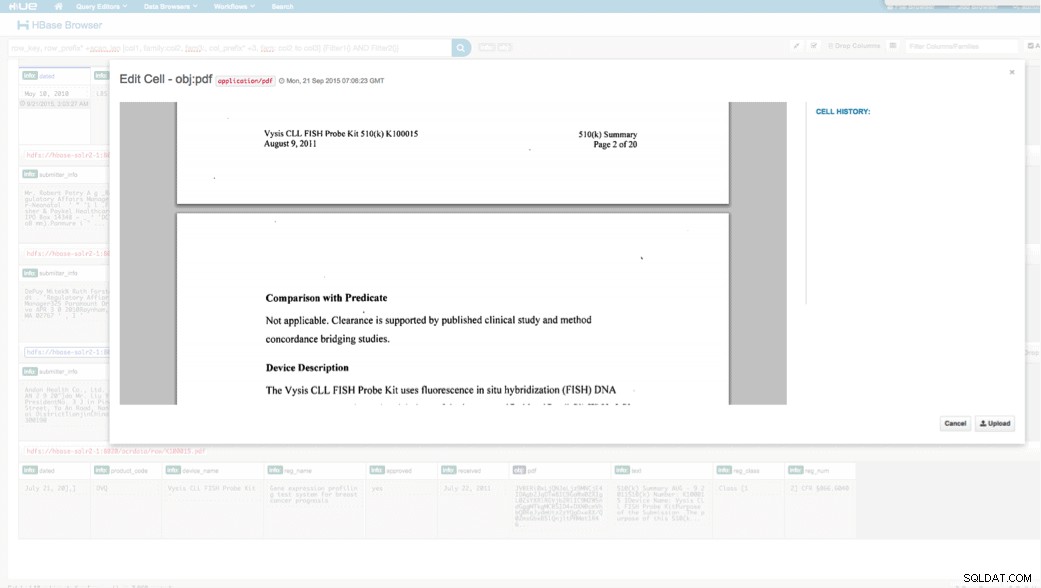
Accesso tramite HUE
Possiamo visualizzare i dati caricati tramite il browser HBase in HUE. Una cosa grandiosa di HUE è che è in grado di rilevare i binari per PDF e renderli quando si fa clic su.
Di seguito è riportato un'istantanea della vista dei campi analizzati nelle righe HBase e anche una vista di rendering di uno degli oggetti PDF archiviati come MOB nella famiglia di colonne obj.
Conclusione
In questo post, abbiamo dimostrato come utilizzare le tecnologie open source standard per eseguire l'OCR su documenti scansionati utilizzando un programma Spark scalabile, archiviando in HBase per un rapido recupero e indicizzando le informazioni estratte in Solr. Dovrebbe essere evidente che:
- Dato il formato della specifica del messaggio, possiamo estrarre campi e coppie di valori e renderli ricercabili tramite Solr.
- Questi campi dei dati possono soddisfare i requisiti IDMP per rendere elettronici i dati legacy, che entreranno in vigore il prossimo anno.
- I campi e le immagini non elaborate possono essere mantenuti in HBase e accessibili tramite API standard.
Se hai bisogno di elaborare documenti scansionati e combinare i dati con varie altre origini nella tua azienda, considera l'utilizzo di una combinazione di Spark, HBase, Solr, insieme a Tesseract e Leptonica. Potrebbe farti risparmiare una notevole quantità di tempo e denaro!
Jeff Shmain è Senior Solution Architect presso Cloudera. Vanta oltre 16 anni di esperienza nel settore finanziario con una profonda conoscenza del trading di titoli, del rischio e delle normative. Negli ultimi anni ha lavorato a varie implementazioni di casi d'uso presso 8 delle 10 maggiori banche di investimento del mondo.
Vartika Singh è Senior Solution Consultant presso Cloudera. Ha oltre 12 anni di esperienza nell'apprendimento automatico applicato e nello sviluppo di software.